
일 시
| 2019년 2월 18일(월)- 26일(화)
장 소
| 서울의대 동창회관 3층 가천홀
등록 바로가기
주 관
|
서울의대 정보의학실, 한국보건복지인력개발원, 시스템 바이오 정보의학 연구센터
(SBI-NCRC)
주 최 |
서울대학교병원 유전체 임상 정보분석 훈련센터
제16차 Genome Data Analysis Workshop을 개최하며
|
안녕하십니까?
제16회 유전체 데이터 분석 웍샵에 여러분을 초대합니다.
올해 웍샵은 정밀의료 인력양성
사업과 협력으로 진행됩니다. 사회 각층의 유전체 데이터 분석과 바이오정보의학 전문인력 양성에 대한 관심이 커졌음을 실감합니다.
새로운 사실을 찾아가는 호기심과 열정도 중요하지만, 그 결과물이 참으로 사람에게 실제적 도움을 줄 수 있는지에 대한 질문과 그에
대한 답변의 책임이 커져갑니다. 저희 유전체 데이터 분석 웍샵은 실사구시적 참된 가치를 생산하기 위한 재료와 방법론과 관점을
제공하기를 지향합니다.
벌써 16번 째를 맞은 웍샵을 통해 확장된 7일간의 웍샵으로 바이오-정보-의학이 진정으로 융합하여 실사의학적 가치를 창출하는 계기를 마련하고자 하오니 많은 관심과 참여를 부탁드립니다. 감사합니다.
2019 년 2월, 서울의대 김 주 한
|
|
|
제15차 GDA Workshop:
2018년 8월 27일~31일, 서울의대
제15차 웍>샵에서는 다음과 같은 실습
모듈이 추가되었다.
(1) Public genetic characterization of a large panel of human cancer cell lines
(2) Informatics for Cancer immunotherapy
제1차 GDA Workshop:
2011년 8월 22일~26일, 서울의대
제2차 GDA Workshop:
2012년 2월 20일~24일, 서울의대
제2차 웍샵에서는 다음과 같은 새로운 실습모듈 3개가 추가 되었다.
(1) micro-RNA 데이터 분석
(2) 개인유전체 해석: Personal Genome Interpretation
(3) 암유전체/희귀질환유전체 데이터 분석
제3차 GDA Workshop:
2012년 8월 20일~24일, 서울의대
제3차 웍샵에서는 다음과 같은 2개의 실습모듈이 추가되었다.
(1) Family-based 엑솜시퀀싱 분석
(2) TCGA (The Cancer Genome Atlas) 데이터 분석
제4차 GDA Workshop:
2013년 2월 18일~22일, 서울의대
제4차 웍샵에서는 다음과 같은 2개의 실습모듈이 추가되었다.
(1) eQTL 데이터 분석
(2) PheWAS & EWAS 데이터 분석
제5차 GDA Workshop:
2013년 8월 26일~30일, 서울의대
제5차 웍샵에서는 다음과 같은 새로운 실습모듈 3개가 추가 되었다.
(1) 시퀀스 레벨 전사체 분석: Isoforms, Alternative Splicing, RNA-editing, and Fusion Gene
(2) 개인유전체 해석을 위한 지식/데이터기반 자원 소개와 유전적 위험 예측 분석
(3) Post-GWAS: EMR 데이터와 질병 연관 분석
제6차 GDA Workshop:
2014년 2월 24일~28일, 서울의대
제6차 웍샵에서는 다음과 같은 새로운 실습모듈 2개가 추가 되었다.
(1) Human Genome Data Analysis using ENCODE
(2) Cancer Genome Data Analysis using TCGA
제7차 GDA Workshop:
2014년 8월 25일~29일, 서울의대
제7차 웍샵에서는 다음과 같은 새로운 실습모듈 2개가 추가 되었다.
(1) Functional Annotation of Sequence Variants
(2) Expression Profiling and Alleleic Status using TCGA RNA-seq Data
제8차 GDA Workshop:
2015년 2월 23일~27일, 서울의대
제8차 웍샵에서는 다음과 같은 새로운 실습모듈 1개가 추가 되었다.
(1) The Final Release of the 1000 Genomes Project Data (2,504 samples) Usage
제9차 GDA Workshop:
2015년 8월 24일~28일, 서울의대
제9차 웍샵에서는 다음과 같은 새로운 실습모듈 2개가 추가 되었다.
(1) Rare, common and disease variant interpretation
(2) Clinical applications of Genetics and Genomics
제10차 GDA Workshop:
2016년 2월 22일~26일, 서울의대
제10차 웍샵에서는 다음과 같은 새로운 실습모듈 2개가 추가 되었다.
(1) The Dominant Regulatory microRNAs and Epigenetic Modulators
(2) Genomic Understanding of Common and Complex Diseases
제11차 GDA Workshop:
2016년 8월 22일~26일, 서울의대
제11차 웍샵에서는 다음과 같은 실습모듈이 추가되었다.
(1) Advanced methods and algorithms for genetic and genomic data analysis
(2) Alternative Splicing and Polyadenylation Analysis
제12차 GDA Workshop:
2017년 2월 20일~24일, 서울의대
제12차 웍샵에서는 다음과 같은 실습모듈이 추가되었다.
(1) Somatic mutation/CNV detection and Mutual exclusivity and coverage
(2) Statistical tests for rare variant association
제13차 GDA Workshop:
2017년 8월 21일~25일, 서울의대
제13차 웍샵에서는 다음과 같은 실습모듈이 추가되었다.
(1) Pathway and Network Analysis of Cancer Genomes
제15차 GDA Workshop:
2018년 8월 27일~31일, 서울의대
제15차 웍샵에서는 다음과 같은 실습
모듈이 추가되었다.
(1) Public genetic characterization of a large panel of human cancer cell lines
(2) Informatics for Cancer immunotherapy
|

실습서
"유전체 데이터 분석" 출간
|
|
■ 준비사항
실습을 위한 참가자 전원 개인 노트북 컴퓨터 준비
R (http://www.r-project.org/), Bioconductor(http://www.bioconductor.org/)
실습실 환경상 인터넷이 느릴수 있으므로 강의 1주일전 안내되는 필요 파일을 필히 다운로드 해오시기바랍니다.
강좌 일정
|
강좌일정은 주최측의 사정에 따라 변경될 수 있습니다.
|
DAY 1: Translational Bioinformatics: Public Bio Big Data
|
|
2월 18일(월)
|
|
시간
|
주 제
|
강 사 |
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
Translational Bioinformatics: Thousands of Public Data Analysis
|
김주한 교수
|
|
9:50 ~ 10:40
|
The Cancer Genome Atlas (TCGA) Project and Cancer Genome Research
- TCGA Introduction
- TCGA Data and Scientific Findings
- Impact of TCGA and Future
|
김태민 교수
(카톨릭의대)
|
|
10:50 ~ 12:10
|
실 습 I: TCGA Somatic Mutation Landscape
Find Significantly Mutated Genes
Identify Driver Groups of Mutations
|
유승원, 김기태
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
Public genetic characterization of a large panel of human cancer cell lines
- CCLE & GDSC Introduction
- Identify drug sensitivity in cell line data
- Gene Expression in cell line data
|
이수연 박사
(기초과학연구원)
|
|
14:10 ~ 15:30
|
실 습 II: Cell line Data at GDSC & CCLE
Gene expression in human cancer cell lines
Comparing Drug sensitivity by cell lines
Epigenetic Regulation of Gene Expression
|
이정훈, 유승원
|
|
15:40 ~ 16:30
|
Public Genome and Exome Data and Human Genome Diversity
- Applications of The 1000 Genomes Project data
- The genome Aggregation Database (gnomAD)
- Mutation Frequencies in Diverse Populations
|
이계화 교수
(서울대병원)
|
|
16:40 ~ 18:00
|
실 습 III: The 1000 Genomes Project Browser
Allele Frequency and Genotypic Frequency Calculation
Identification of Rare and Deleterious Mutations
|
박유미, 정문경
|
|
DAY 2: Methodology and Application for Next Generation Sequencing
|
|
2월 19일(화)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
Methodology and Application for Next Generation Sequencing |
김주한 교수
|
|
9:50 ~ 10:40
|
NGS Platforms and Applications
- Current NGS Platforms
- NGS Data Formats
- NGS Data Analysis Technologies
- NGS Applications
|
김지훈 박사
(녹십자지놈)
|
|
10:50 ~ 12:10
|
실 습 I: NGS Data Processing
NGS Data Format Converting
NGS Visualization Tools
|
박유미, 안세환
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
NGS Data Analysis
- Sequence Alignment Algorithms
- Whole Genome and Exome Data Analysis
- Variation Detection and Reference Genome
|
최무림 교수
(서울의대)
|
|
14:10 ~ 15:30
|
실 습 II: Exome Sequencing Alignment
SNP and Indel Identification
Variation Filtering
|
임영균, 한봄
|
|
15:40 ~ 16:30
|
Exome Sequencing Analysis
- Exome Sequencing Data
- Study Design and Workflow
- Exome Sequencing of Familial Disease
|
한미령 교수
(고려의대)
|
|
16:40 ~ 18:00
|
실 습 III: Trio-Exome-Sequencing Data Analysis
Known Variant Filtering
Detection of Disease-causing Variations
Disease Gene Prioritization
|
유경훈, 박유미
|
|
DAY 3: Cancer Genome Bioinformatics
|
|
2월 20일(수)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
Cancer Genome Bioinformatics
|
김주한 교수
|
|
9:50 ~ 10:40
|
Cancer Genome Bioinformatics
- Cancer Genome Analysis
- Genomic Rearrangement and Copy Number
- Somatic Variant detection/ Gene Fusion Analysis
- Survival analysis
|
송영수 교수
(한양의대)
|
|
10:50 ~ 12:10
|
실 습 I: Somatic Variant Detection
Survival Analysis
Resources for Cancer Research: cBioPortal, COSMIC, CCLE
|
이우승, 임영균
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
Advanced Cancer Genome Data Analysis
- Different types of variation in cancer
- Matched normal and tumor pair data analysis
- Cancer clonality and sequencing depth
- Visualizing cancer genomics data
|
정제균 박사
(삼성유전체연구소)
|
|
14:10 ~ 15:30
|
실 습 II: Somatic mutation/CNV detection and genome data processing
Mutual exclusivity and coverage
Visualization of cancer study
|
김기태, 채정환
|
|
15:40 ~ 16:30
|
Informatics for Cancer immunotherapy
- Genomics in cancer immunotherapy
- Tumor mutation burden(TMD) and neoantigen
- Gene expression and immune cells
|
김태형 박사
(테라젠)
|
|
16:40 ~ 18:00
|
실 습 III: Tumor mutation burden(TMB) calculation
Neoantigen Prediction
Identification of marker for immune cells
|
임영균, 권호식
|
|
DAY 4: NGS-based Rare and Common Disease Variant Analysis
|
|
2월 21일(목)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
NGS-based Rare and Common Disease Variant Analysis
|
김주한 교수
|
|
9:50 ~ 10:40
|
Rare and common disease variant analysis using NGS
- CVCD vs. RVCD: synthetic associations
- Variant prioritization strategy
- Interpretation of deleterious variants
- Gene-centric approaches
|
김주한 교수
(서울의대)
|
|
10:50 ~ 12:10
|
실 습 I: Functional effects of variant (CADD, SIFT, PolyPhen2)
Gene-wise analysis using gene scoring system (RVIS, GDI)
|
권호식, 윤선민
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
Advanced methods and algorithms for genetic association analysis
- Statistical methods for genomic association tests
- HLA imputation and fine-mapping
- Analysis of clinical heterogeneity and pleiotropy
|
한 범 교수
(서울의대)
|
|
14:10 ~ 15:30
|
실 습 II: Statistical tests for association (Chi-square, Fisher's exact test and Odds ratio
Rare variant association tests (Burden tests, SKAT, SKAT-O)
|
이정훈, 최선
|
|
15:40 ~ 16:30
|
NGS Applications of Clinical Genetics and Genomics
- Understanding genetic risk prediction
- Genetic counseling
- Incidental findings from genomic sequencing data
|
이계화 교수
(서울대병원)
|
|
16:40 ~ 18:00
|
실 습 III: Genetic Risk Prediction methods
ACMG recommended genes annotation
Resources
for personal genome interpretation(ClinVar, HGMD, PheGeni, SNPedia,
PhenoDB)
|
윤선민, 정문경
|
|
DAY 5: RNA-Seq Data Analysis
|
|
2월 22일(금)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
RNA-Seq Data Analysis
|
김주한 교수
|
|
9:50 ~ 10:40
|
RNA-Seq Data Analysis
- Read Alignment Methods
- Expression Quantification Strategy
- Differentially Expressed Genes Identification
|
강근수 교수
(단국대)
|
|
10:50 ~ 12:10
|
실 습 I: RNA-seq Data Preprocessing with old(tophat, cufflinks) & recent(hisat2, stringtie) Tuxudeo Pipeline
|
이우승, 채정환
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
Sequence-level Transcriptome Analysis
- Single Cell RNA-Seq data analysis
- Alternative Polyadenylation Analysis
- RNA Editing Analysis
|
박지연 박사
(가톨릭의대)
|
|
14:10 ~ 15:30
|
실 습 II: Single Cell RNA-Seq Analysis
RNA-DNA Difference (RDD) Analysis
RNA Editing Site Annotation
|
유경훈, 최 선
|
|
15:40 ~ 16:30
|
Alternative Splicing and Non-coding RNAs
- RNA-Seq data analysis
- Alternative Splicing Events
- Non-coding RNA Characterization
|
이영희 교수
(유타대)
|
|
16:40 ~ 18:00
|
실 습 III: Alternative Splicing analysis
Non-coding RNA chracterization and analysis
|
김주연, 유경훈
|
|
DAY 6: Advanced Transcriptome Data Analysis
|
|
2월 25일(월)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
Advanced Transcriptome Data Analysis
|
김주한 교수
|
|
9:50 ~ 10:40
|
Biological Interpretation of Transcriptome Data
- Gene Ontology & Pathway Analysis
- Gene Set Enrichment Analysis
- Prognostic Subgroup Prediction
|
김주한 교수
(서울의대)
|
|
10:50 ~ 12:10
|
실 습 I: Gene Set Enrichment Analysis
Cox-PH, Log Rank Test
|
김주연, 윤선민
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
Non-coding RNA Analysis
- miRNA-seq Expression Profile Analysis
- Novel Transcript Discovery
- Non-coding RNA Characterization
|
정제균 박사
(삼성유전체연구소)
|
|
14:10 ~ 15:30
|
실 습 II: RNA-Seq Gene Expression Analysis
miRNA Sequencing Data Process
miRNA Expression Profiling
|
안세환, 임영균
|
|
15:40 ~ 16:30
|
Pathway and Network Analysis
- Characteristics of Biological Network
- Cancer Gene Network Construction and Clustering
- Identification of Aberrant Pathway in Cancers
|
한현욱 교수
(차의대)
|
|
16:40 ~ 18:00
|
실 습 III: Pathway analysis of TCGA using PARADIGM methods
Network Visualization in Cancers
|
김기태, 이정훈
|
|
DAY 7: Noncoding regulatory sequence and Epigenome analysis
|
|
2월 26일(화)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치 |
|
9:30 ~ 9:50
|
Noncoding regulatory sequence and Epigenome analysis
|
김주한 교수
|
|
9:50 ~ 10:40
|
Functional interpretation of noncoding regulatory variants
- Epigenomic Annotation of Noncoding Variant
- Understanding Gene Regulatory Mechanisms
- Regulatory Network Analysis with Transcriptomics
|
남석우 교수
(가톨릭의대)
|
|
10:50 ~ 12:10
|
실 습 I: ENCODE Data at UCSC Genome Browser
DNA Mutations in Genome Level
Functional Methylation/Histone Modification in Epigenetic Level
Epigenetic Regulation of Gene Expression
|
윤선민, 김주연
|
|
12:10 ~ 13:10
|
중 식
|
|
13:10 ~ 14:00
|
Motif and Regulatory Sequence Analysis
- Sequence Motif Analysis
- Genome Sequence Analysis
- Genome Browser
- Metagenome analysis
|
박지연 박사
(가톨릭의대)
|
|
14:10 ~ 15:30
|
실 습 II: TF Target Prediction for Metagenomes
Phylogenetic Analysis (ClustalW & TreeView)
UCSC Genome Browser
|
권호식, 김기태
|
|
15:40 ~ 16:30
|
Epigenome Data Analysis
- Epigenetic Mechanisms
- DNA Methylation Analysis
- Histone Modification Analysis
- Discovery of Epigenetic Biomarkers
|
김 선 교수
(서울대)
|
|
16:40 ~ 18:00
|
실 습 III: Epigenome Tools & Databases
Visualization of DNA Methylation Data
Identification of Methylated Genes
|
유승원, 이우승
|
|
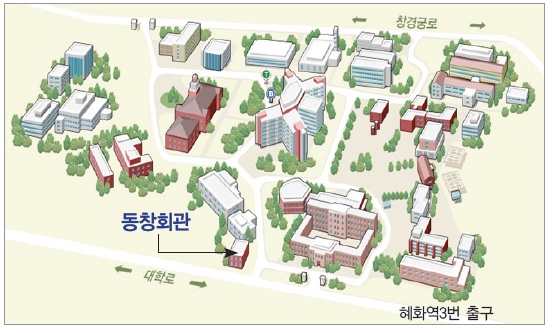
오시는 길
|
교육장소 |
서울의대 동창회관 3층 가천홀, 서울시 종로구 대학로 103
교 통 | 지하철 4호선 혜화역 3번 출구
|
|






