|
강좌일정은 주최측의 사정에 따라 변경될 수 있습니다.
|
DAY 1: Advanced NGS Data Analysis
|
|
2월 20일(월)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치
|
|
9:30 ~ 10:20
|
Epigenome Data Analysis
- Epigenetic Mechanisms
- DNA Methylation Analysis
- Histone Modification Analysis
- Discovery of Epigenetic Biomarkers
|
정제균 교수
(차의대)
|
|
10:30 ~ 12:00
|
실 습 I: ENCODE and UCSC Genome Browser
Methylation data & Histone modification data
Chip-Seq data Analysis for regulatory SNP
|
최선 (6장)
|
|
12:00 ~ 13:00
|
중 식
|
|
13:00 ~ 13:50
|
Pathway and Network Analysis
- Characteristics of Biological Network
- Cancer Gene Network Construction and Clustering
- Identification of Aberrant Pathway in Cancers
|
한현욱 교수
(차의대)
|
|
14:00 ~ 15:30
|
실 습 II: Statistical tests for association
Rare variant association tests (Burden tests, SKAT, SKAT-O)
HLA imputation
|
조원일 (25장)
|
15:40 ~ 16:30
|
Multiomics and Precision Medicine
- Genome, Transcriptome, and Proteom Integration
- miRNA and Exosome Analysis
- Application for Precision Medicine
|
류성호 교수
(순천향대학교)
|
|
16:40 ~ 18:10
|
실 습 III: Genomics Characterization of Human Cancer Cell Lines
Gene expression in human cancer cell line
Comparing Drug sensitivity by cell lines
Drug-gene databases
|
부은경 (7장)
|
|
18:10 ~ 18:30
|
질의응답 및 마무리
|
|
DAY 2: Methodology and
Application for Next Generation Sequencing
|
|
2월 21일(화)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치
|
|
9:30 ~ 10:20
|
NGS Platforms and Applications
- Current NGS Platforms
- NGS Data Formats
- NGS Data Analysis Technologies
- NGS Applications
|
인용호 박사
(싸이퍼롬)
|
|
10:30 ~ 12:00
|
실 습 I: NGS Data Formats
NGS Data
Format Converting
NGS
Visualization Tools
|
이시은 (9장)
|
|
12:00 ~ 13:00
|
중 식
|
|
13:00 ~ 13:50
|
NGS Data Analysis
- Sequence Alignment Algorithms
- Whole Genome and Exome Data Analysis
- Variation Detection and Reference Genome
|
최무림 교수
(서울의대)
|
|
14:00 ~ 15:30
|
실 습 II: Sequence Variant Calling and Annotation
Exome
Sequencing Alignment
SNP,
INDEL Identification and Filtering
|
윤미선 (10장)
|
|
15:40 ~ 16:30
|
Informatics for Cancer immunotherapy
- Genomics in cancer immunotherapy
- Tumor mutation burden(TMD) and neoantigen
- Gene expression and immune cells
|
장현
교수
(카톨릭관동의대)
|
|
16:40 ~ 18:10
|
실 습 III: Informatics for Cancer immunotherapy
Variant annotation by VEP
Neoantigen Prediction
Identification of marker for immune cells
|
임영균 (17장)
|
|
18:10 ~ 18:30
|
질의응답 및 마무리
|
|
DAY 3: Cancer Genome and Multi-omic Bioinformatics
|
|
2월 23일(목)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치
|
|
9:30 ~ 10:20
|
The Cancer Genome Atlas (TCGA) Project and Cancer Genome Research
- TCGA Introduction
- TCGA Data and Scientific Findings
- Impact of TCGA and Future
|
홍동완 교수
(카톨릭의대)
|
|
10:30 ~ 12:00
|
실 습 I: Cancer Somatic Mutation Analysis
Somatic
Variant Detection
Survival
Analysis
Resources
for Cancer Research
|
전예진 (13,14장)
|
|
12:00 ~ 13:00
|
중 식
|
|
13:00 ~ 13:50
|
Exome Sequencing Analysis
- Exome Sequencing Data
- Study Design and Workflow
- Exome Sequencing of Familial Disease
|
김태형 박사
(테라젠)
|
|
14:00 ~ 15:30
|
실 습 II: Annotating Functional Effect of Sequence
Variants
Introduction
to ANNOVAR
Use
of GENECODE, Ensembl, BioMart, etc
|
배소정 (11장)
|
|
15:40 ~ 16:30
|
Advanced Cancer Genome Data Analysis
- Different types of variation in cancer
- Matched normal and tumor pair data analysis
- Cancer clonality and sequencing depth
- Visualizing cancer genomics data
|
이수연 교수
(아주대)
|
|
16:40 ~ 18:10
|
실 습 III: Genomic Rearrangement, CNV, and Mutual
exclusivity
Mutual
exclusivity and coverage
Visualization
of cancer study
|
유준기 (16장)
|
|
18:10 ~ 18:30
|
질의응답 및 마무리
|
|
DAY 4: RNA-Seq and
Transcriptome Data Analysis
|
|
2월 24일(금)
|
|
시간
|
주 제
|
강 사
|
|
8:30 ~ 9:30
|
등록 및 사전 프로그램 설치
|
|
9:30 ~ 10:20
|
RNA-Seq Data Analysis
- Read Alignment Methods
- Expression Quantification Strategy
- Differentially Expressed Genes Identification
|
김기태 교수
(서울대)
|
|
10:30 ~ 12:00
|
실 습 I: RNA-Seq Transcriptome Basic Data Analysis
Old Tuxedo
Pipeline(tophat, cufflinks)
New Tuxedo
Pipeline(hisat2, stringtie)
DEG analysis
|
권호식 (20장)
|
|
12:00 ~ 13:00
|
중 식
|
|
13:00 ~ 13:50
|
Sequence-level Transcriptome Analysis
- Alternative Splicing Events
- Alternative Polyadenylation Analysis
- RNA Editing Analysis
|
박지연 박사
(카톨릭의대)
|
|
14:00 ~ 15:30
|
실 습 II: Alternative Splicing Identification
RNA-DNA
Difference (RDD) Analysis
RNA
Editing Site Annotation
|
조민아 (21장)
|
|
15:40 ~ 16:30
|
Non-coding RNA Analysis
- miRNA-seq Expression Profile Analysis
- Novel Transcript Discovery
- Non-coding RNA Characterization
|
이영희 교수
(서울대)
|
|
16:40 ~ 18:10
|
실 습 III: Micro-RNA and non-coding RNA Data
Analysis
RNA-Seq
Gene Expression Analysis
miRNA
Sequencing Data Process
miRNA
Expression Profiling
|
안세환 (22장)
|
|
18:10 ~ 18:30
|
질의응답 및 마무리
|
■
등 록
등록인원
: 하루 강좌 당
42명 내외
참 가 비
: 하루 강좌 당 참가비
등록방법
:
http://www.snubi.org/workshop/the23rdgda/registration.html
| |
2월 6일 이전
|
2월 7일 이후
|
|
공공/대학/정부
|
100,000
|
120,000
|
|
일반(기업등)
|
120,000
|
160,000
|
*
한 사람이 3개의 강좌까지 선택 수강 가능
교재는 각자 구매를 해 오셔야 하며, 교재명은 범문에듀케이션 간
유전체 데이터 분석
II편 - NGS편
입니다.
강의대상: 대규모 유전체 데이터 분석과 응용에
관심 있는 BT, IT, 혹은 의학 분야 전공자
환불규정: 2월 6일 이전 취소: 전액 반환
2월 7일 ~ 2월 19일 취소: 50% 환불
2월 20일 이후: 환불 불가
등록 신청 후 3일 (공휴일, 주말 제외) 이내에 아래의 계좌로 입금해 주십시오.
신한은행: 100-030-071195 예금주: 한국정보의학인증의관리위원회
■ 준비사항
실습을 위한 참가자 전원 개인 노트북 컴퓨터 준비(최소 쿼드코어 이상 CPU, RAM
16GB 이상이여야 원할한 진행 가능)
Apple의 M1, M2 탑재된 PC는 호환성 문제로 실습 프로그램이 작동 안 할 수도 있으니, 다른 PC를 지참 부탁드립니다.
R (http://www.r-project.org/),
Bioconductor(http://www.bioconductor.org/)
실습실 환경 상 인터넷이 느릴수 있으므로, 강의 1주일 전 안내되는 필요
파일을 필히 다운로드 부탁드립니다.
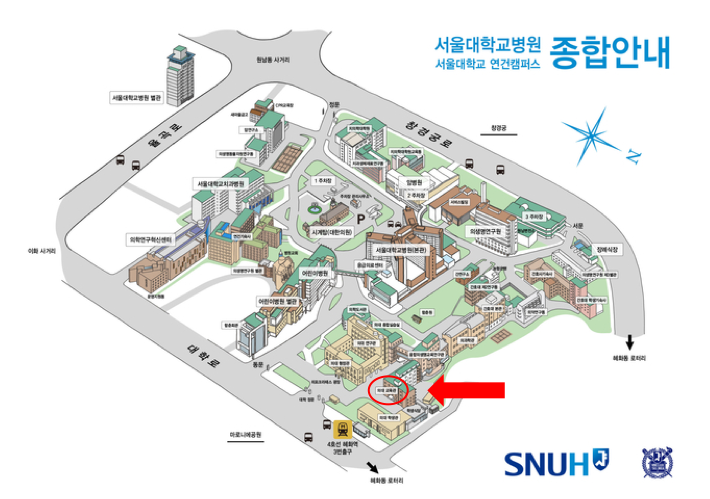
오시는 길
|
교육장소 |
서울대학교 의과대학 교육관 117호, 서울시 종로구 대학로 103
교 통 |
지하철 4호선 혜화역 3번 출구
|
|





